新疆理化所在RNA N6-甲基腺苷修饰位点识别方面取得进展
N6-甲基腺苷(N6-methyladenosine, m6A)是最常见的RNA修饰之一,是一种关键的表观遗传因子信号,对剪接调节、mRNA稳定性、翻译效率和表观遗传调控有着深远的影响。尽管已经开发了各种计算模型来准确识别潜在的m6A修饰位点,但大多缺乏通过共识知识来解释修饰位点识别过程的能力。
为了克服这一问题,中国科学院新疆理化技术研究所科研团队提出了一种深度学习模型,即M6A-DCR,发现RNA序列上的共识区域以促进m6A修饰位点的可解释识别。M6A-DCR首先通过整合核苷酸的特定位置和类型来构建每个RNA序列的实例图,并将共识区域的发现转换为聚集所有实例图的图聚类问题。之后,进一步采用基序感知的图重构优化过程,学习输入RNA序列的高质量嵌入,从而实现m6A修饰位点的端到端识别。通过与几种最先进的识别模型进行比较,实验结果证明了所提出的方法的优越性能。通过不同物种、不同组织的交叉验证分析,进一步验证了M6A-DCR鉴定结果与物种间进化关系的一致性以及相关效应蛋白与所发现共识区域的潜在关联(图1)。这表明考虑共识区域使该模型能够在基序水平上做出可解释的识别。
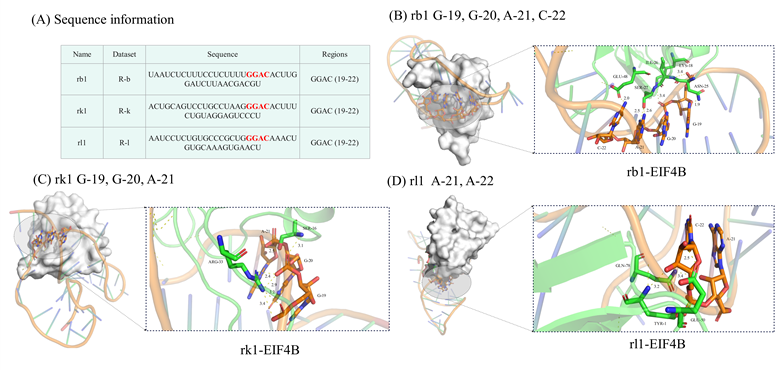
图1. 效应蛋白与识别的共识区域对接细节
该研究为RNA m6A修饰位点的计算鉴定提供了有价值的见解。相关研究成果以《Discovering consensus regions for interpretable identification of RNA N6-methyladenosine modification sites via graph contrastive clustering》为题发表在生物信息领域国际顶级期刊IEEE Journal of Biomedical and Health Informatics (Volume: 28, Issue: 4, Pages: 2362 - 2372, April 2024),并被选为封面文章(图2)。
该研究工作得到了国家自然科学基金、新疆维吾尔自治区自然科学基金、中国科学院率先行动人才计划以及CAS西部之光-西部交叉团队项目的资助。新疆理化所为唯一完成单位,胡伦研究员为通讯作者,2022级博士李国栋为第一作者。IEEE Journal of Biomedical and Health Informatics是中国科学院1区TOP期刊,最新影响因子为7.7。
文章链接:https://ieeexplore.ieee.org/document/10413556

图2. 文章封面